本篇文章给大家分享的是有关如何自Python中使用Entrez库筛选并下载PubMed文献,小编觉得挺实用的,因此分享给大家学习,希望大家阅读完这篇文章后可以有所收获,话不多说,跟着小编一起来看看吧。
任务:快速高效从PubMed上下载满足条件的文献PMID,标题(TI)摘要(AB)。
PubMed官网https://pubmed.ncbi.nlm.nih.gov
此处有几种选择可以达到目的:
(1)官网上匹配筛选条件(注:匹配快速,但是下载下来的数量受到限制,每次只能下载10000条数据,甚至更少)。
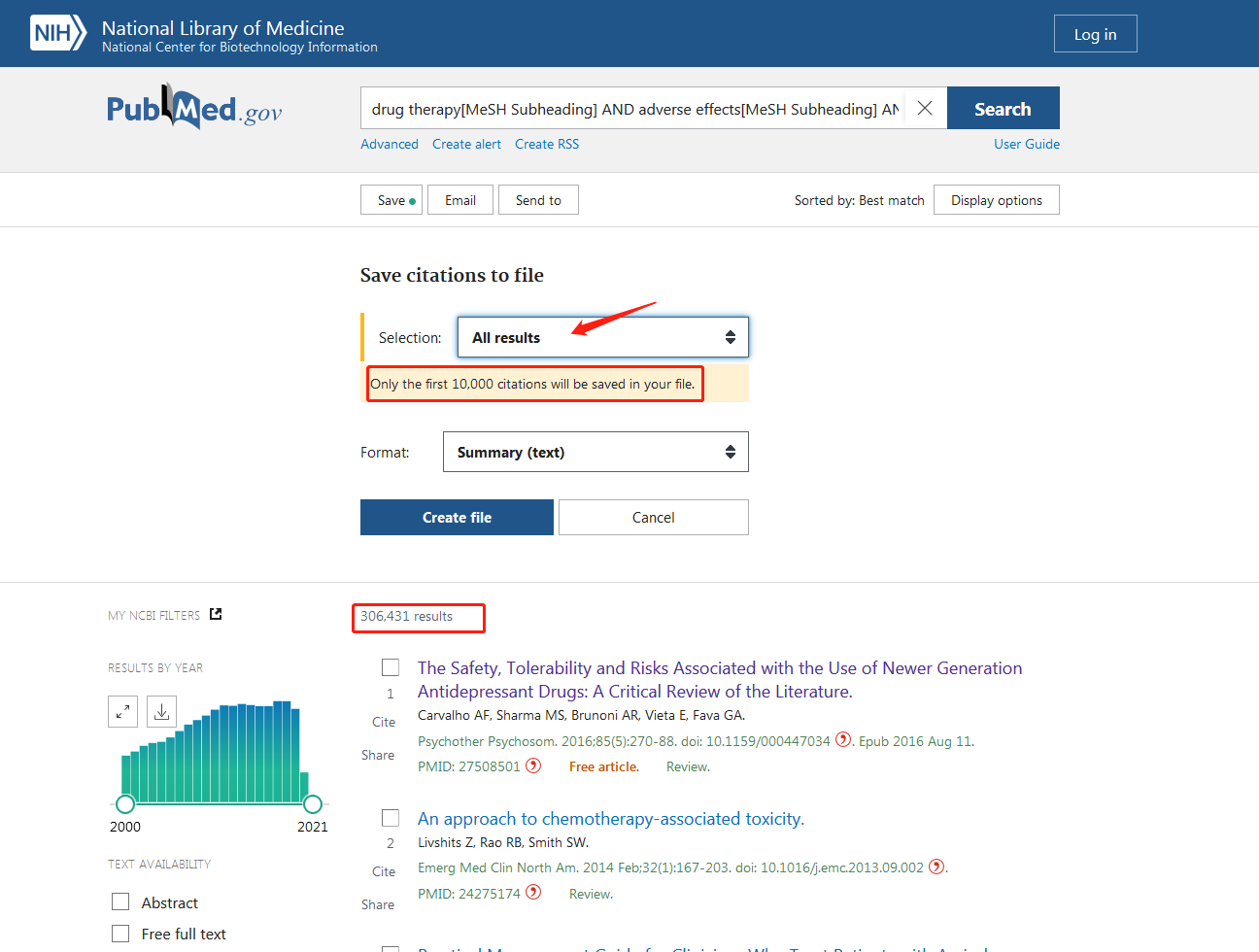
,可以看的到,我需要的数据是有三十多万条,但是每次只能下载10000条,那我岂不是要手动n次. .很明显,在大批量下载文献的情况下,官网不是很友好。
(2) R语言有个R包,叫做easyPubMed,这里我也给大家贴上学习指南(https://cran.r-project.org/web/packages/easyPubMed/vignettes/getting_started_with_easyPubMed.html)
由于我不喜欢用R写代码,所以我写一半还是换了Python,熟练R的小伙伴可以自行根据指南走通需求。
(3)重量级库来了,Python自带的生物包中提供的Entrez检索库,简直就是我的救星,以下是我的代码:
注:可以在生物包中,生物的安装请移步https://www.cnblogs.com/xiaolan-Lin/p/14023147.html
进口numpy np 从生物进口Medline, Entrez #一般是通过BioPython的Bio.Entrez模块访问可以 从进口计数器集合 可以。电子邮件=?此处写你自己在官网注册的邮箱账号)“;#应用自己的账号访问NCBI数据库 #此处需将服务器协议指定为1.0,否则会出现报错.http.client。IncompleteRead: IncompleteRead(0字节读) 1.0 #服务器http协议,而python的是1.1,解决办法就是指定客户端http协议版本 进口http.client http.client.HTTPConnection。_http_vsn=10 http.client.HTTPConnection。_http_vsn_str=& # 39; HTTP/1.0 & # 39; “““ 可以是一个检索系统,可以用其访问NCBI数据库,比如说PubMed、基因库、地理等。 获得有关全球多溴二苯醚的所有文献的PubMed id “““ # handle_0=Entrez.esearch (db=皃ubmed"词=耙┪镏瘟?副标题)和不利影响(副标题)和人类(网格计算)“,retmax=306431) handle_0=Entrez.esearch (db=皃ubmed"词=耙┪镏瘟?网状副标题)和不利影响(网状副标题)和人类(网格计算)和(2000/01/01[日期-出版]:2021/12/31[出版日期-])“, ptyp=癛eview" usehistory=皔" retmax=306431) 记录=Entrez.read (handle_0) #获取检索条件的所有文献 idlist=记录(“IdList") #提取出文献id 打印(“总:“记录[“Count"]) No_Papers=len (idlist) #共306431篇文献2000 - 01 - 01:2021 - 12 - 31所示 webenv=记录[& # 39;WebEnv& # 39;】 query_key=记录[& # 39;QueryKey& # 39;】 总=No_Papers 一步=1300 打印(“结果项目:“总) 张开(“。/Data_PubMed/PBDE1.txt", & # 39; w # 39;) f: 开始的范围(0总步骤): 打印(“下载记录% i %我;%(+ 1开始,int(开始+一步))) handle_1=Entrez.efetch (db=皃ubmed" retstart=开始,rettype=癿edline", retmode=皌ext", retmax=一步,webenv=webenv query_key=query_key) #获取上述所有文献的PubMed id 记录=Medline.parse (handle_1)=列表记录(记录)#将迭代器转换至列表(列表) 在np.arange指数(len(记录): id=记录(指数). get (“PMID",,, # 63;“) title=记录(指数). get (“TI",,, # 63;“) title=title.replace (& # 39; [& # 39; & # 39; & # 39;) .replace(& # 39;] # 39;公司& # 39;& # 39;)#若提取的标题出现[]。符号,则去除 文摘=记录(指数). get (“AB",,, # 63;“) f.write (id) f.write (“\ n") f.write(标题) f.write (“\ n") f.write(抽象) f.write (“\ n")
话不多说,结果跑出来了我真的很快乐~

最后的结果是存放在txt文件中,大伙儿根据自己的需求改变代码所需字段啊。
现在我来解释一下,我贴上的这串代码的实现原理,首先是通过Entrez检索到符合我筛选条件的文献,里边的限制条件包括了几个词汇匹配以及时间限制,时间我限制在了2000年1月1日到2021年的12月31日(这里的时间我选用的是日期,出版时间选取日期,完成日期,修改还是日期——出版其实还是有争议的,大家自行考虑选取)。