本篇内容主要讲解”如何通过环球数码创意遗产档案下载TCGA原始数据”,感兴趣的朋友不妨来看看。本文介绍的方法操作简单快捷,实用性强。下面就让小编来带大家学习”如何通过环球数码创意遗产档案下载TCGA原始数据”吧!
在2016年之前,TCGA项目的相关结果文件存放在CGhub TCGA和数据协调中心简称DCC提供TCGA的数据门户中,当时的结果是以hg19或者hg18为参考得到的。
在DCC中,将数据划分为了3个等级. level 1代笔原始的,未经处理的数据的,比如芯片下机数据;二级代表处理的中间结果,比如测序深度对应的假发文件;三级代表处理完成后的最终结果,比如基因的定量结果。
2016年之后,CGhub和DCC相继关闭,所有的数据统一迁移到现在用的环球数码创意数据库,而且通过环球数码创意的管道将原有的结果转换为hg38参考基因组版本。目前在环球数码创意中检索到的结果都是经过了环球数码创意管道处理过后的,从这里也可以看的出,迁移到hg38是一个大的趋势。
当然目前使用hg19的还是挺多的,如果你需要基于hg19版本TCGA的数据,在环球数码创意中也可以找到。其环球数码创意中实的数据可以分为以下两个部分
<李>
环球数码创意遗产档案
在R包<代码> TCGAbiolinks>

第一部分就是默认使用的基于hg38版本的数据,第二部分则是对原始TCGA的结果的一个存储,通过环球数码创意首页的环球数码创意应用<代码> CDC遗产档案>
https://portal.gdc.cancer.gov/legacy-archive
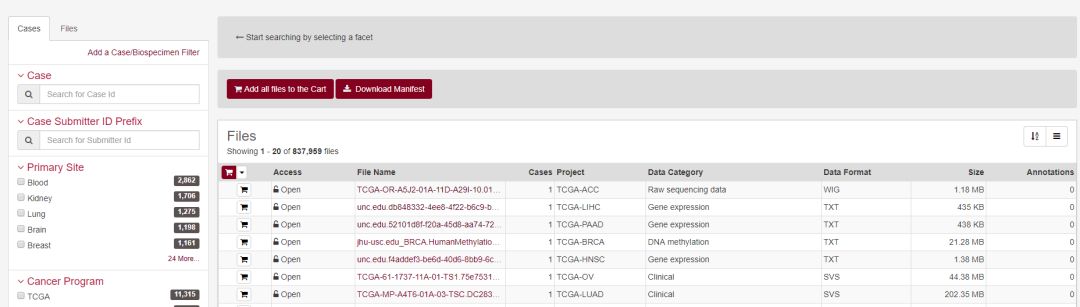
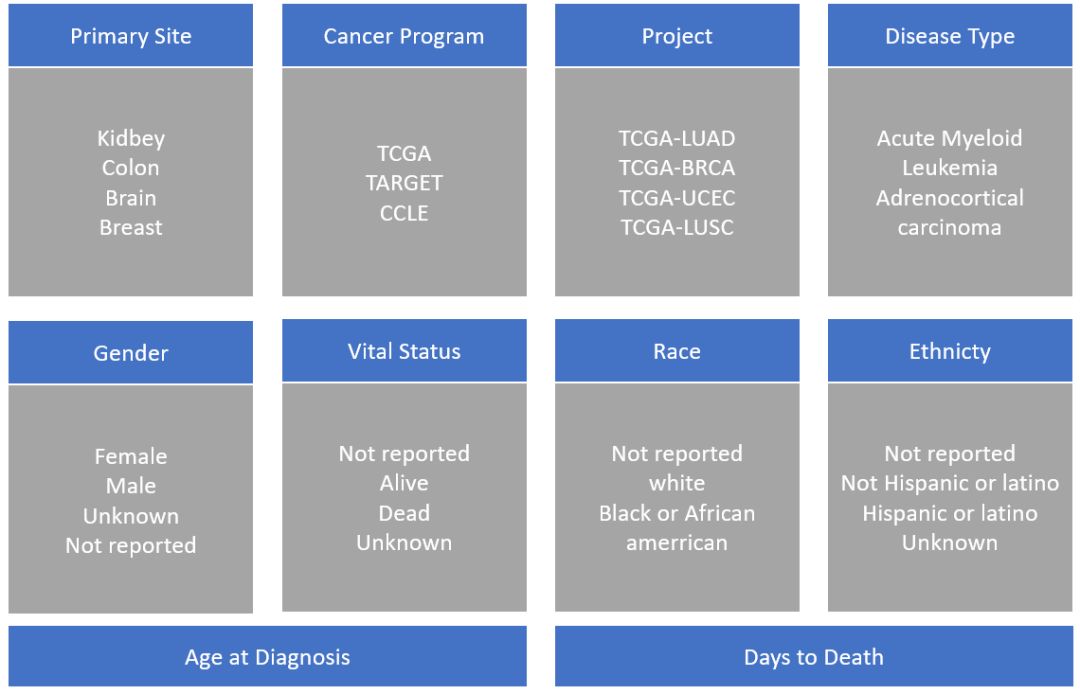
在左侧的面板可以根据相关属性对病例和文件进行筛选,病例相关的属性如下
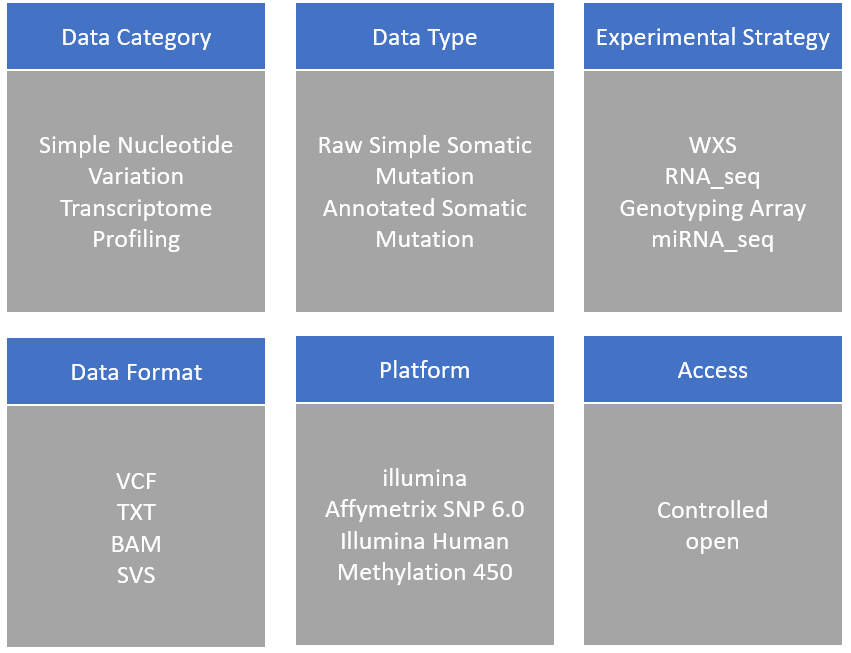
文件相关的属性如下
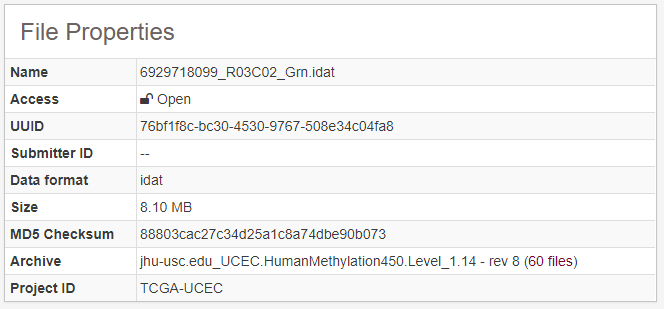
数据的下载方式和前面文章中介绍的相同,这里不赘述,从文件名称可以看到对应的水平,不同级别的文件示意如下
1。使
通过<代码>数据类型> 进行筛选,得到芯片的原始数据,示意如下
2。,二级
通过<代码>数据类型为<代码>报道假发进行筛选,得到比对的测序深度数据,示意如下
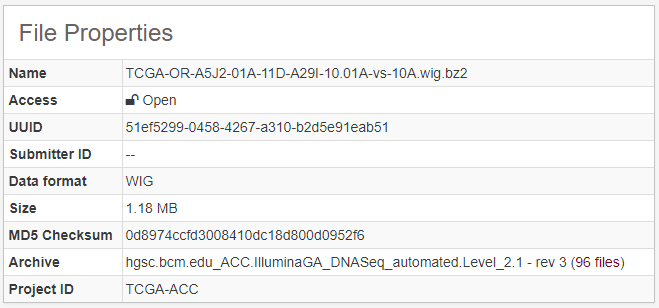
3。level3
通过<代码>数据类型为<代码> microrna基因量化进行筛选,得到microrna的表达定量数据,示意如下
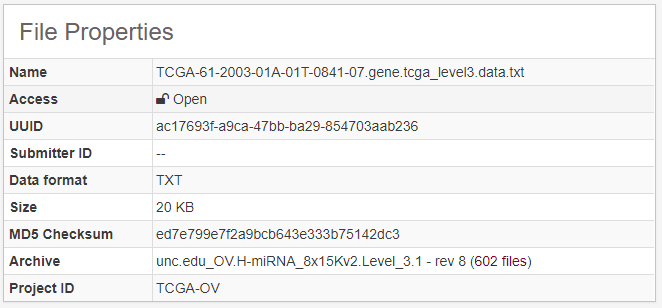
通过环球数码创意遗留存档,可以找到基于hg19的数据结果文件,但是由于相关的网站已经关闭,无法确认该数据分析的pipieline等细节信息,所以需要谨慎使用。
<节> <节> <节> <节> <节>
到此,相信大家对“如何通过环球数码创意遗产档案下载TCGA原始数据”有了更深的了解,不妨来实际操作一番吧!这里是网站,更多相关内容可以进入相关频道进行查询,关注我们,继续学习!
环球数码创意遗产如何通过存档下载TCGA原始数据





